• Неконтролируемый анализ: метод, который не учитывает какие-либо известные группы выборки, является исследовательским. Примерами неконтролируемых методов являются – метод главных компонент (PCA), метод проекций на скрытые структуры (PLS), а также канонический анализ корреляции (CCA).
• Контролируемый анализ: метод включает вектор, указывающий на принадлежность класса в каждой выборки. Цель его состоит в том, чтобы различать выборочные группы и выполнять прогнозирование для класса выборки. Примерами контролируемых методов являются дискриминантный анализ проекций на скрытые компоненты (PLS-DA), анализ интеграции данных для обнаружения маркеров с использованием скрытых компонентов (DIAB), а также многомерный интегративный метод определения воспроизводимых сигнатур в независимых экспериментах на разных платформах (MINT).
Перечень широко используемых методов mixOmics, которые будут подробно описаны в соответствующих главах ниже, за исключением CCA и MINT, можно представить следующей таблицей типов и объема данных, который они могут обрабатывать:
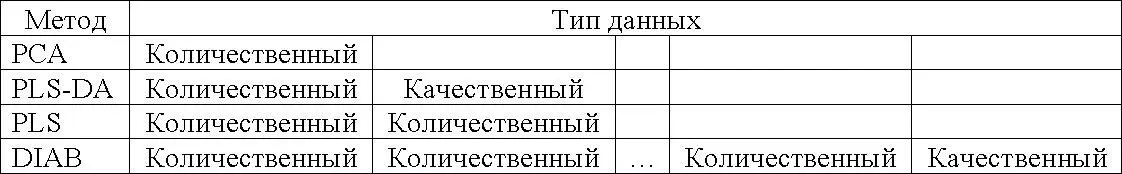
Методы, реализованные в mixOmics, подробно описаны в разных публикациях, обширный список которых постоянно пополняется и может быть найден в открытых источниках.
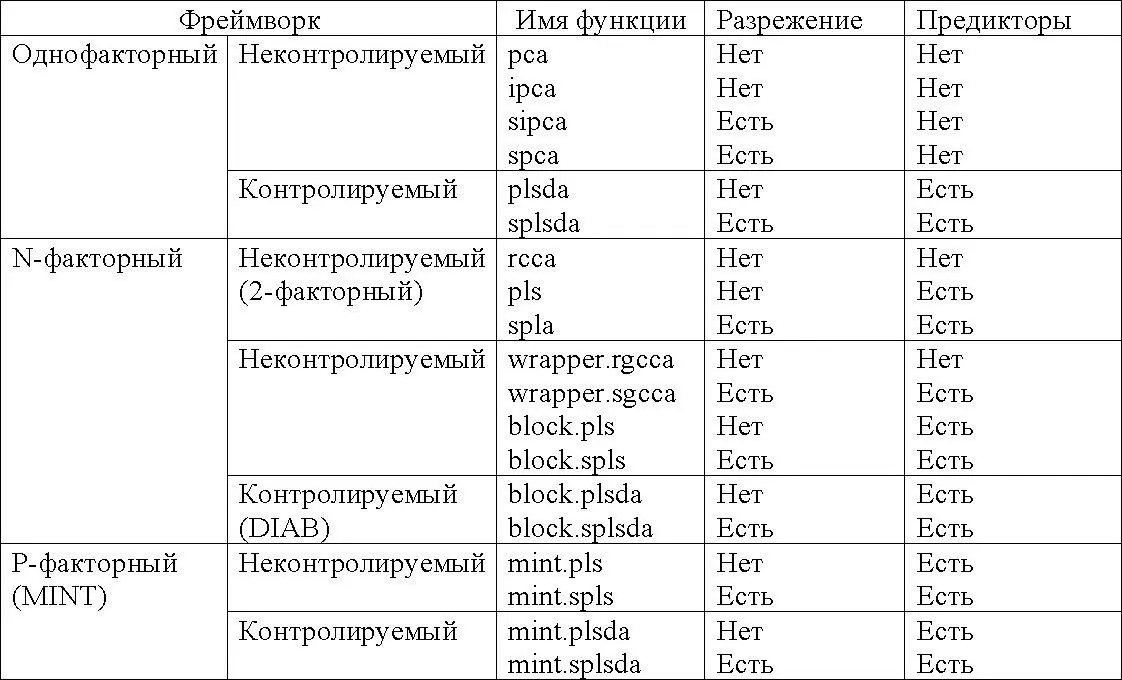
В следующей таблице приведён список методов mixOmics, наличие разрежения в которой указывает на методы, предполагающие осуществление выбора переменных:
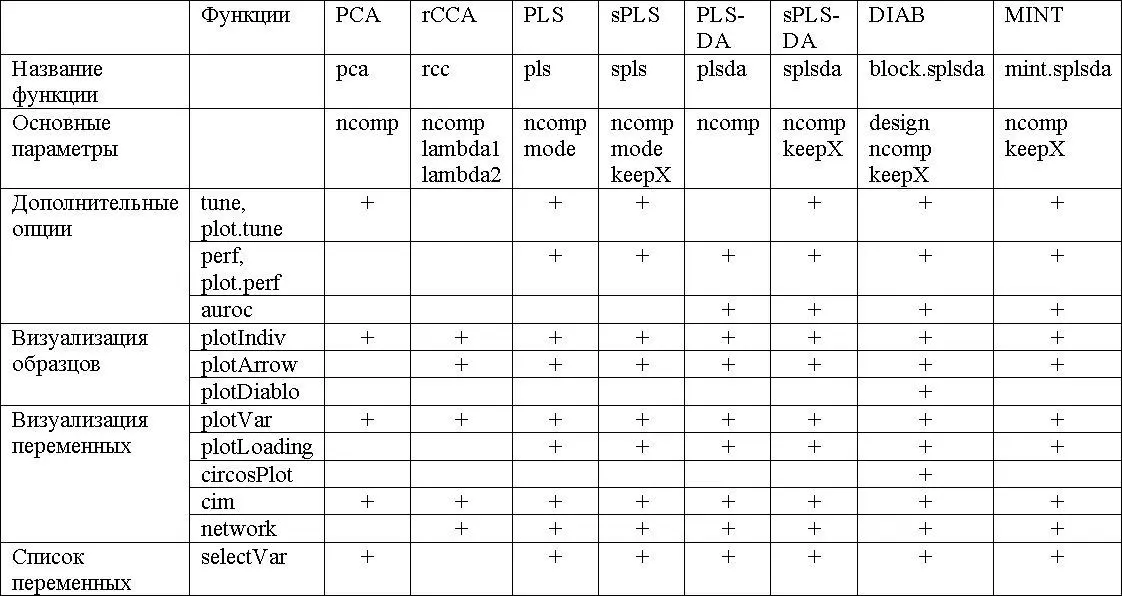
Основные функции и параметры каждого метода сведены в следующей таблице:
Каждый раздел, посвященный описанию того или иного метода, излагается по следующему плану:
1. Тип педагогического вопроса, на который нужно ответить.
2. Краткое описание иллюстративного набора данных.
3. Принцип метода.
4. Быстрый запуск метода с основными функциями и аргументами.
5. Чтобы идти дальше: настраиваемые опции, дополнительные графические построения и настройки параметров.
6. Вопросы и ответы.
Как путь в тысячу миль начинается с первого шаг, так и использование любого пакета R начинается с его установки. Во-первых, можно скачать последнюю версию mixOmics от Bioconductor следующей командой:
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("mixOmics")
Кроме того, можно установить последнюю версию пакета с GitHub, но для этого понадобится предварительная установка пакета remotes:
BiocManager::install("remotes")
BiocManager::install("mixOmicsTeam/mixOmics")
Пакет mixOmics напрямую импортирует следующие пакеты: igraph, rgl, ellipse, corpcor, RColorBrewer, plyr, parallel, dplyr, tidyr, reshape2, methods, matrixStats, rARPACK, gridExtra. Если возникнут затруднения при установке пакета rgl, то нужно будет дополнительно установить программное обеспечение X'quartz.
Загрузить установленный пакет можно следующей командой:
library(mixOmics)
Убедитесь, что при загрузке пакета не возникло ошибки, особенно для упомянутой выше библиотеки rgl. В примерах, которые будут приведены далее, используются данные, являющиеся частью пакета mixOmics. Чтобы загрузить свои собственные данные, проверьте установлен ли рабочий каталог, а затем считайте данные из формата .txt или .csv, либо с помощью пункта меню импортирования данных в RStudio, либо через одну из следующих командных строк:
# из файла csv
data <���– read.csv("имя_файла.csv", row.names = 1, header = TRUE)
# из файла txt
data <���– read.table("имя_файла.txt", header = TRUE)
Для получения более подробной информации о аргументах, используемых для настройки параметров этих функций, введите ?read.csv или ?read.table в консоли R.
Каждый анализ должен выполняться в следующем порядке:
1. Запустите выбранный метод анализа.
2. Выполните графическое представление образцов.
3. Выполните графическое представление переменных.
Затем используйте критическое мышление и дополнительные функции инструментов визуализации, чтобы разобраться в полученных данных. Некоторые из вспомогательных инструментов будут описаны в следующих главах.
Например, для анализа основных компонентов сначала загружаем данные:
My_table <���– structure(list(Класс = c("7а", "7а", "7а", "7а", "7а", "7а", "7а", "7а", "7а",
"7а", "7б", "7б", "7б", "7б", "7б", "7б", "7б", "7б", "7б", "7б", "эталон", "отстающий"),
`Фимилия Имя` = c("Иванов Иван", "Петров Петр", "Сидоров Сидор", "Егоров Егор",
Читать дальше














