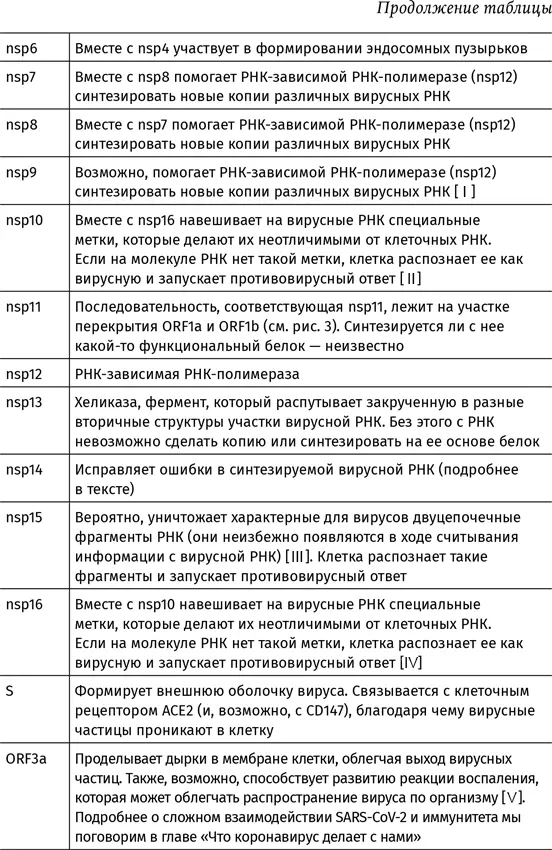
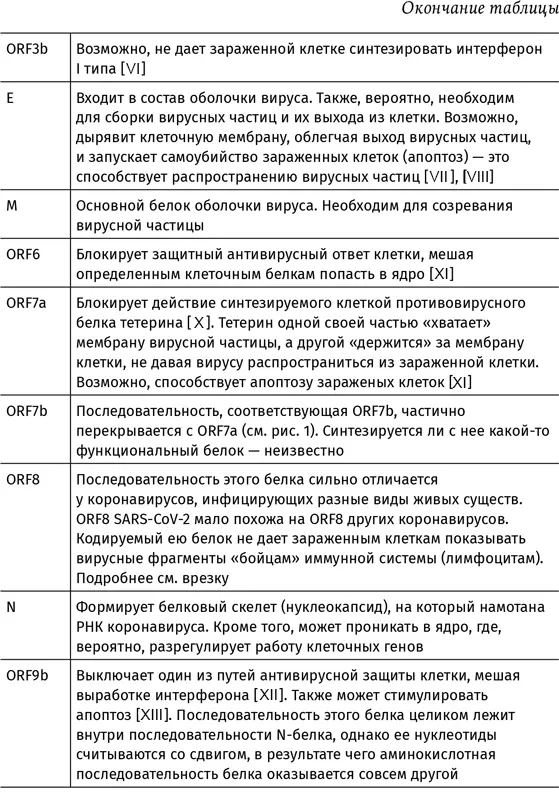
1 ...6 7 8 10 11 12 ...134 Полный список белков коронавируса SARS-CoV-2 и их функции описаны в таблице 1. Аббревиатурой nsp обозначают неструктурные белки (от английского non-structural proteins ). К ним относятся сервисные белки вируса вроде РНК-зависимой РНК-полимеразы и белков, обеспечивающих сборку вирусных частиц, а также nsp, выключающие собственный метаболизм клетки и мешающие ей позвать на помощь иммунную систему. Остальные белки составляют каркас вирусной частицы.

В геноме SARS-CoV-2 видны еще три последовательности, с которых, теоретически, могли бы считываться белки — ORF9c, ORF10 и ORF14. Но действительно ли они «работают», или это просто артефакт — неизвестно.
[I] M.-P. Egloff et al. , «The severe acute respiratory syndrome-coronavirus replicative protein nsp9 is a single-stranded RNA-binding subunit unique in the RNA virus world», Proc. Natl. Acad. Sci. , vol. 101, no. 11, pp. 3792–3796, Mar. 2004.
[II] Y. Wang et al. , «Coronavirus nsp10/nsp16 Methyltransferase Can Be Targeted by nsp10-Derived Peptide In Vitro and In Vivo To Reduce Replication and Pathogenesis», J. Virol. , vol. 89, no. 16, pp. 8416 LP-8427, Aug. 2015.
[III] Там же.
[IV] V. D. Menachery, K. Debbink, and R. S. Baric, «Coronavirus non-structural protein 16: Evasion, attenuation, and possible treatments», Virus Res. , vol. 194, pp. 191–199, Dec. 2014.
[V] K. Siu et al. , «Severe acute respiratory syndrome Coronavirus ORF3a protein activates the NLRP3 inflammasome by promoting TRAF3-dependent ubiquitination of ASC», FASEB J. , vol. 33, no. 8, pp. 8865–8877, Aug. 2019.
[VI] Y. Konno et al. , «SARS-CoV-2 ORF3b is a potent interferon antagonist whose activity is further increased by a naturally occurring elongation variant», bioRxiv , p. 2020.05.11.088179, Jan. 2020.
[VII] D. Schoeman and B. C. Fielding, «Coronavirus envelope protein: current knowledge», Virol. J. , vol. 16, no. 1, p. 69, Dec. 2019.
[VIII] «UniProtKB — P59637 (VEMP_CVHSA). Envelope small membrane protein», UniProt . [Online]. Available: https://www.uniprot.org/uniprot/P59637. [Accessed: 14-Aug-2020].
[XI] M. Frieman, B. Yount, M. Heise, S. A. Kopecky-Bromberg, P. Palese, and R. S. Baric, «Severe Acute Respiratory Syndrome Coronavirus ORF6 Antagonizes STAT1 Function by Sequestering Nuclear Import Factors on the Rough Endoplasmic Reticulum/Golgi Membrane», J. Virol. , vol. 81, no. 18, pp. 9812–9824, Sep. 2007.
[X] J. K. Taylor et al. , «Severe Acute Respiratory Syndrome Coronavirus ORF7a Inhibits Bone Marrow Stromal Antigen 2 Virion Tethering through a Novel Mechanism of Glycosylation Interference», J. Virol. , vol. 89, no. 23, pp. 11820–11833, Dec. 2015.
[XI] S. R. Schaecher and A. Pekosz, «SARS Coronavirus Accessory Gene Expression and Function», in Molecular Biology of the SARS-Coronavirus , Berlin, Heidelberg: Springer Berlin Heidelberg, 2010, pp. 153–166.
[XII] C.-S. Shi et al. , «SARS-Coronavirus Open Reading Frame-9b Suppresses Innate Immunity by Targeting Mitochondria and the MAVS/TRAF3/TRAF6 Signalosome», J. Immunol. , vol. 193, no. 6, pp. 3080–3089, Sep. 2014.
[XIII] C.-S. Shi, N. R. Nabar, N.-N. Huang, and J. H. Kehrl, «SARS-Coronavirus Open Reading Frame-8b triggers intracellular stress pathways and activates NLRP3 inflammasomes», Cell Death Discov. , vol. 5, no. 1, p. 101, Dec. 2019.
Глядя на таблицу 1, можно подумать, что мы уже очень много знаем о белках SARS-CoV-2. На самом деле это не так. Большинство представленных здесь сведений получены учеными, исследовавшими вирус SARS. Геномные последовательности SARS и SARS-CoV-2 сходны на 79 % [13] K. B. Anand, S. Karade, S. Sen, and R. M. Gupta, «SARS-CoV-2: Camazotz’s Curse», Med. J. Armed Forces India , vol. 76, no. 2, pp. 136–141, Apr. 2020.
, и большинство белков практически неотличимы друг от друга. Но очевидно, с каждым месяцем исследователи будут лучше разбираться в подрывной деятельности нового коронавируса. Так, предварительный анализ взаимодействия белков SARS-CoV-2 с белками клетки показал, что даже безобидные структурные белки теоретически могут вмешиваться в те или иные иммунные процессы [14] D. E. Gordon et al. , «A SARS-CoV-2 protein interaction map reveals targets for drug repurposing», Nature , vol. 583, no. 7816, pp. 459–468, Jul. 2020.
. Чем лучше мы будем понимать, как именно коронавирус воздействует на организм, тем больше у нас будет потенциальных слабых мест, на которые можно нацеливать лекарства.
Об одном белке нового коронавируса нужно упомянуть отдельно. Это nsp14 — фермент, умеющий исправлять ошибки, допущенные при копировании вирусных РНК. В первой главе мы говорили, что РНК-содержащие вирусы отличаются повышенной склонностью мутировать. Одна из причин — помимо нестабильности самой молекулы РНК — это крайне неряшливая работа РНК-зависимой РНК-полимеразы, фермента, который синтезирует новые копии вирусной РНК. По сравнению, например, с ДНК-полимеразами, которые копируют молекулы ДНК, она допускает колоссально много ошибок {8} 8 Столь существенная разница связана не с тем, что ДНК-полимеразы не допускают ошибок: допускают, но умеют самостоятельно исправлять их.
. Так много, что это угрожает выживанию вируса: если не исправлять их, очень быстро последовательность изменится настолько, что на ее основе нельзя будет синтезировать ни одного работающего белка. Поэтому коронавирусы обзавелись ферментом-пруфридером (от английского proofreading , то есть правка текста корректором, в ходе которой он исправляет ошибки). Он «видит» неправильно вставленные РНК-полимеразой нуклеотиды и вырезает их. После этого полимераза вновь пытается вставить в это место нуклеотид — и так как ошибается она, все же, не каждый раз, со второй попытки опечатка часто исчезает. Впервые исправляющий ошибки белок обнаружили у SARS, и, похоже, это уникальная придумка именно коронавирусов. По крайней мере, ни у каких других РНК-вирусов такого фермента не найдено.
Читать дальше
Конец ознакомительного отрывка
Купить книгу







![Арсений Сухоницкий - Эмгед Гард / Мне достался божий сценарий и я не знаю, что с ним делать. Том 2 [СИ]](/books/390105/arsenij-suhonickij-emged-gard-mne-dostalsya-bozhij-thumb.webp)
![Арсений Сухоницкий - Эмгед Гард / Мне достался божий сценарий и я не знаю, что с ним делать [СИ]](/books/390106/arsenij-suhonickij-emged-gard-mne-dostalsya-bozhij-thumb.webp)
![Илья Мартынов - Мозг. Как он устроен и что с ним делать [litres]](/books/405254/ilya-martynov-mozg-kak-on-ustroen-i-chto-s-nim-del-thumb.webp)



