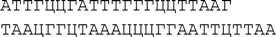Изобретение Сэнгером в середине 1970-х годов метода секвенирования ДНК оказалось важнейшей вехой на пути создания базы данных о последовательностях ДНК всевозможных организмов. Но как раз в отношении создания таких баз данных это изобретение опередило свое время. Ведь тогда еще не был доступен Интернет, а без Интернета создание и использование базы данных о последовательностях ДНК практически немыслимо. Так что первые десять лет накопление знаний о различных геномах шло медленно, хотя и были сделаны важнейшие открытия, о которых мы говорили выше в этой главе и еще будем говорить в главе 6. Кроме Интернета, важнейшим изобретением, резко ускорившим и упростившим создание геномных баз данных, был метод полимеразной цепной реакции (ПЦР), который позволил амплифицировать, т. е. многократно приумножать любые выбранные участки генома. Но метод ПЦР заслуживает особого разговора, собственно, с него началась биотехнологическая революция, и мы о нем подробно поговорим в главе 10.
Метод Сэнгера позволяет секвенировать куски ДНК, содержащие около 1000 нуклеотидов, но они, конечно, гораздо короче геномной ДНК. Как же секвенировать целый геном, содержащий, в случае человеческого генома, 3 миллиарда нуклеотидов? Понятно, что геномную ДНК надо нарезать на короткие куски. Слава богу, у нас есть такой сверхточный инструмент: рестиктазы (см. главу 4). Итак, используя какую-нибудь рестриктазу или смесь двух рестриктаз, если хотим, чтобы куски были покороче, нарезаем ДНК на куски (рис. 19). Прекрасно, теперь можно прочесть каждый кусок методом Сэнгера. Но постойте, для метода Сэнгера нужен праймер. Откуда же нам знать, какой праймер использовать, ведь мы еще ничего не знаем о последовательности кусков? Как же быть? А очень просто. Ведь после действия рестриктазы у фрагментов, как правило, образуются «липкие концы». Например, после разрезания ДНК рестриктазой EcoRI образуются два взаимно комплементарных конца:

Но эти концы одинаковые, так что если мы сделаем на ДНК-синтезаторе такую искусственную молекулу:
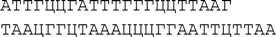
то она прилипнет к обоим концам, образовавшимся под действием рестриктазы. Правда, в обоих случаях между нашей синтетической молекулой, которая называются адаптером, и куском неизвестной пока ДНК имеются два однонитевых разрыва, но это не беда: они легко залечиваются ферментом ДНК-лигазой. Теперь все наши фрагменты, полученные после нарезания геномной ДНК, оказываются снабженными по концам прекрасно известной нам последовательностью, ведь мы ее сами выдумали, когда делали дизайн адаптеров: все 20 нуклеотидов слева от концевого Г в верхней цепи адаптера я выдумал сам, совершенно произвольно. Так что теперь нет никакой проблемы с дизайном праймеров для чтения последовательностей кусков геномной ДНК методом Сэнгера. Снабженные адаптером фрагменты разделяются с помощью гель-электрофореза или каким-нибудь другим способом (рис. 19), а затем секвенируются.
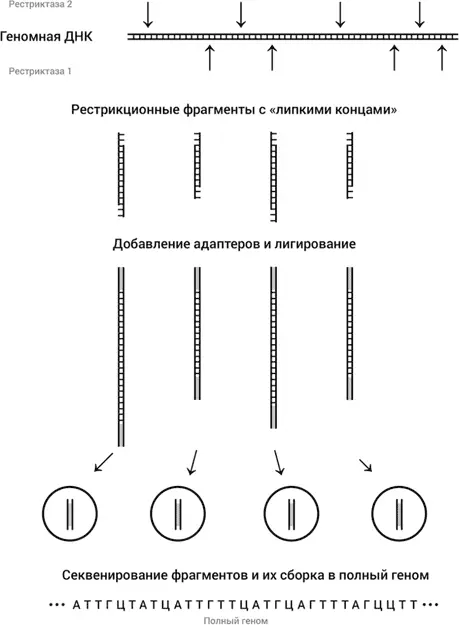
Рис. 19.Секвенирование генома. Вся геномная ДНК подвергается разрезанию на фрагменты рестриктазой (то же самое повторяется с использованием другой рестриктазы, чтобы в дальнейшем на последней стадии провести сборку всей последовательности по перекрывающимся участкам фрагментов, полученных при разрезании разными рестриктазами). Рестрикционные фрагменты соединяются с синтетическими адаптерами, как объяснено в тексте, с использованием «липких» концов, создаваемых рестриктазой. Затем фрагменты разделяются, и каждый фрагмент отдельно секвенируется, после чего следует сборка всего генома
Итак, мы секвенировали все куски, на которые была порезана геномная ДНК рестриктазой EcoRI. Дело в шляпе? Не тут-то было! Мы же не знаем, в каком порядке расположены куски вдоль генома. Как их теперь правильно собрать? К сожалению, нет другого способа, как повторить все сначала, используя другую рестриктазу. Тогда мы получим другое разрезание и по перекрывающимся участкам сможем узнать, какой кусок, полученный с помощью первой рестриктазы, следует за куском, полученным с помощью второй рестриктазы (рис. 19). Конечно, такая сборка полной последовательности делается компьютером. Но повторное секвенирование так и так надо делать, чтобы избежать случайных ошибок, ведь, как бы ни была хороша ДНК-полимераза, она редко, но ошибается. В реальности, чтобы получить последовательность генома с очень малым количеством ошибок, всю процедуру повторяют 10 раз.
Читать дальше
Конец ознакомительного отрывка
Купить книгу